
|
| Halsbandparkiet, Ring-necked Parakeet, Psittacula krameri |
Happy New Year for all blog visitors!
 |
| Feasting on peanuts. Left: female. Right: male. |
Gene-centrism (defined as genes create and control the organism) is bad biology, not because it is an ideology or a philosophy, but because it is an incorrect description of what happens in a cell. Gene-centrism is embedded in the language in which biologists usually write and think about DNA. In order to eliminate gene-centrism, we need to take a fresh look at the role of DNA in cells and organisms. Criticisms such as "DNA is not the book of life" and "DNA is not a blueprint" are too vague and ineffective.
Here is an example of DNA centrism:
"Central to this are enhancers and promoters, DNA sequences that dictate the location, timing, and intensity of gene expression."
from: A tighter grip on gene expression, Science 3 Jul 2025.
Please note: "dictate"! and "timing"! . Especially, 'timing' is mysterious. How do DNA sequences dictate timing? I asked the author. No reply. I think this is a sloppy language caused by a mindless DNA-centric view. In general, the question is not asked: Does the genome control the cell? Or does the cell control the genome? Since 'the genome controls the cell' seems to be the default position, it is not a well-argued position. Therefore, it is better to refer to 'causes' than 'reasons' for gene-centrism. [4]
In 1859 there was a big hole in Darwin's theory of evolution. He lacked a theory of heredity. In 1900 Mendelian genetics, and in 1953 the double-helix filled in the hole and transformed biology. Now, biology has a solid theory of heredity. DNA transformed the theory of evolution, too. The opposite of Darwin's theory has become true: DNA quickly became the centerpiece of the theory of evolution. Yet, this centerpiece obstructed a clear view of the actual role of DNA in organisms.
It would be foolish to deny that DNA is an indispensable component of life on earth. But that does not mean that DNA has the power to create and control the development and maintenance of organisms. DNA is not the Uncaused First Cause! DNA is the source, but not the cause! Writing and thinking about DNA should be free of this kind of bias. The supposed arguments for DNA-centrism are not arguments at all, but just sloppy language. Others [2] have criticized DNA-centrism inadequately. In a follow-up post, I will deal with 'the selfish gene'.
De HomeWizard thuisbatterij is een prachtig apparaat, maar is volledig afhankelijk van een stabiele wifi verbinding. Bovendien gebruikt het apparaat alleen de 2,4 GHz wifi frequentie en heeft geen UTP netwerk kabel aansluiting.
Onze Fritz!box router gebruikt zowel 2,4 GHz als 5 GHz wifi, net als onze telefoons. Maar de Homewizard batterij kan niet schakelen tussen die twee frequnties als er veel wifi verkeer is. Hij kan alleen maar de 2,4 GHz band (wifi versie 4) gebruiken. Ik weet niet wat er is veranderd: de software van de router? [2], de software van de batterij? Hebben de buren misschien een extra wifi versterker geïnstalleerd? De Homewizard helpdesk raadde aan om een apart 2,4 GHz thuisnetwerk aan te maken en de Homewizard apparaten daar op aan te sluiten. Dat heb ik gedaan, maar het hielp niet. Een oplossing zou kunnen zijn om wifi versterkers te installeren. Maar dat veroorzaakt volgens mij een wifi-oorlog. Als alle buren wifi versterkers installeren en jij doet het ook, dan heb je niets gewonnen. Wifi versterkers kunnen een bereik van zeker 10 meter hebben. Misschien schaffen de buren wel een set van drie versterkers aan: een complete wifi wapenwedloop! Maar ik heb het anders gedaan. Het is wel een eigenwijze oplossing.
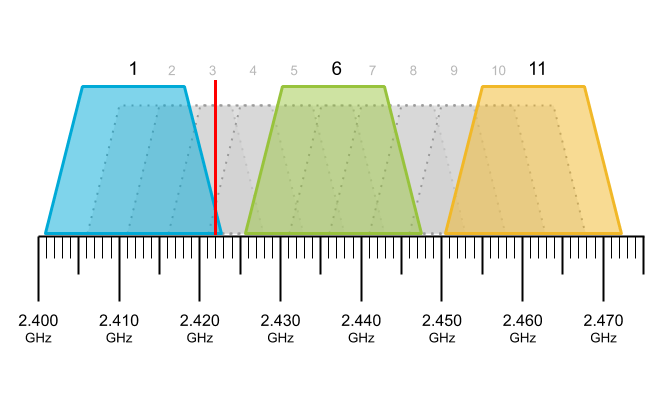 |
| 2,4 GHz. kanaal 3 (= 2,422 GHz) |
Dit plaatje geeft de aanbevolen kanalen 1, 6 en 11 binnen de 2,4 GHz frequentie zoals je die op het internet kunt vinden. De rode streep heb ik toegevoegd. Je kunt vele soortgelijke plaatjes vinden. Dit is een officiële aanbeveling:
"Professionals gebruiken daarom altijd de kanalen 1, 6 en 11. Deze overlappen niet, dus ze veroorzaken onderling geen ruis. Er zijn dus drie ‘schone’ kanalen die ongestoord door elkaar heen kunnen lopen." ( bron )
Veel websites bevelen de kanalen 1, 6 en 11 aan. Google-AI beveelt ze ook aan. Het lijkt wel alsof iedereen elkaar na praat, in plaats van zelf te experimenteren. Op het bovenstaande plaatje zie je dat die frequenties elkaar inderdaad niet overlappen. Dat klopt. Dus als je één van die drie kiest, zit je goed. Zou je denken. Maar bijvoorbeeld mijn smartphone ziet al 13 wifi netwerken. In de router beheersoftware zie je dat alle buren ook kanalen 1, 6, 11 gebruiken. Terug bij af! Dan is het daar alsnog druk. Daarom heb ik kanaal 3 geprobeerd (in rood aangegeven op de bovenstaande figuur). Een gokje. Kanaal 4, of 8, of 9 zou je ook kunnen proberen. Kijk in je eigen router om te zien welke kanalen het drukst zijn. Bij mij werkt kanaal 3 nu ruim drie weken probleemloos [1]. Dat was daarvoor wel anders. De batterij draait er prima mee. Geen last van de buren, want die gebruiken dat 'foute' kanaal niet. De thuisbatterij staat plm. 4 meter van de meterkast inclusief tussenmuur. Een bijkomend voordeel is dat je geen wifi versterkers hoeft aan te schaffen. Het is geen probleem wanneer je de batterij voor een tijdje uitgezet hebt, want hij maakt automatisch weer verbinding zodra je hem weer aanzet.
 |
| Fritz!box Wifi Channel settings |
Hoe kun je een specifiek wifi kanaal instellen? Ga naar de beheersoftware van je router. Zie bovenstaand plaatje: 'Adjust Wi-Fi channel settings'. Vul een channel in onder het kopje 2,4 GHz. Ik heb gekozen voor channel 3.
Het is jammer dat de HomeWizard batterij geen vaste netwerkkabel aansluiting heeft naast de wifi. Je moet mensen de keuze geven. Er bestaat niets betrouwbaarder dan een vaste kabel verbinding. Dat je dat weglaat in het ontwerp van je batterij vind ik onbegrijpelijk. Wifi is altijd storingsgevoelig, en het signaal wordt zwakker met de afstand. Dat nadeel heeft een kabel verbinding niet. Mensen zijn de meest simpele en betrouwbare oplossing vergeten: een netwerkkabel van 5-15 meter door het trappenhuis naar slaapkamer of zolder. Mijn router heeft 3 UTP-netwerkkabel aansluitingen. En die gebruik ik ook.
HomeWizard (de fabrikant).
Wifi versies (wikipedia): Homewizard gebruikt wifi-4: een standaard uit 2009. Waarom een oude versie gebruiken? De nieuwste wifi versie is 7 van 2024, en is backward compatible met oudere versies.
"Internet routers en Wi-Fi-repeaters die meerdere Wi-Fi-netwerken,
zoals bijvoorbeeld de 2,4 GHz- en 5 GHz-frequentieband, tegelijkertijd
beschikbaar stellen, kunnen Wi-Fi-apparaten via band steering
automatisch naar het optimale Wi-Fi-netwerk sturen.
Wi-Fi-apparaten die geschikt zijn voor verbindingen met meerdere
Wi-Fi-netwerken, bepalen zelfstandig met welk Wi-Fi-netwerk zij de
verbinding tot stand brengen. Wi-Fi-apparaten maken echter niet altijd
de optimale keuze. Door band steering kunnen routers en repeaters voor
elk Wi-Fi-apparaat herkennen welk Wi-Fi-netwerk het meest geschikt is
en sturen het apparaat daar automatisch heen."
De apparaten van HomeWizard zijn niet in staat om aan band steering te doen omdat ze alleen op 2,4 GHz werken.
Op 26 Feb 2026 verdween de wifiverbinding van de batterij voor plm 30 sec en herstelde zich automatisch. Oorzaak onbekend. Tot die datum geen rood knipperlicht gezien!

|
Question: is this music? Are the above symbols on a piece of paper music? What do you think? My answer: of course not! It is encoded information. Only a specially trained person is able to read and translate that information into sounds with the help of specially designed devices. On their own, these ink stains are not music.
Question: do the symbols on a piece of paper cause music? Are they the cause of music? My answer: of course not! The ink dots on a piece of paper are not the cause of the sounds we call music. Those symbols are dead and meaningless ink stains in themselves. One needs a person with specific knowledge of how to read and translate the symbols on paper. Furthermore, that person needs a special designed instrument. Do the hands cause the music? Or does the instrument cause the music? Or our ears? Or our brains? It is impossible to point to one cause. It is everything together. It is clear that here we have a chain of causal factors.
DNA centrism
"The basic principle is that if genes were abundantly available in the primordial pond, they could have randomly assembled to form various genomes, each capable of forming an organism." [1]
This is the most concise and extreme expression of DNA-centrism I know of. It is almost a definition of DNA-centrism in the context of the Origin of Life. The statement claims that life started with DNA. All you need is DNA and the organism will develop from it. On the other hand, in biology DNA-centrism means: DNA creates the organism. DNA is the cause of the organism. DNA controls every biological function in the organism. DNA-centrism in the context of Mendelian genetics is almost by definition gene-centrism because Mendelian genetics is not interested in the molecular details of how a gene affects the phenotype of the organism. For Mendelian genetics, the inner workings of an organism are a black box.
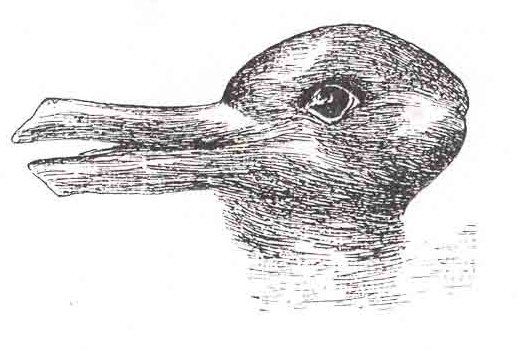
Now, let us ask the same questions about DNA as we did above about the musical notes on a piece of paper: does DNA cause an organism to develop, grow, breath and live? Of course not! Try it yourself: place a complete genome in a physiological saline solution in a Petri dish at 37°C and wait. Nothing will happen! But why? Because this is an unnatural environment? But if DNA has the power to create an organism, why does DNA do that? Apparently, DNA hasn't the power to create an organism. Apparently, the genome has to be in a cell and needs all the machinery to read and translate the information in the genome. Conclusion: it only seems that DNA is the cause of an organism because it is always in the right environment. We always assume the right environment.
Compare this with the musical notes on paper: we are used to associating musical notes on paper with the sounds of music. Change a note (mutation) and the music changes (phenotype). But the musical notes on paper define, but do not create music. The right environment is required to create music [2]. We need a change of perspective to see this.
|
|
Duck-Rabbit illusion (wikipedia) Does DNA control the cell, or does the cell control DNA? |
Genetic disease seem to be a very strong argument against my position and for DNA-centrism. What is the cause of Cystic Fibrosis? The answer: Cystic Fibrosisis is caused by a mutation in the CFTR gene. What is the cause of Huntington's Disease? Answer: HD is caused by a mutation in the Huntingtin gene. What is the cause of Duchenne Muscular Dystrophy? DMD is caused by a mutation of the dystrophin gene. What is the cause of Sickle cell disease? Sickle cell disease is caused by a mutation in the HBB gene. A clear case for DNA-centrism?
No. On closer inspection, it appears that genetic diseases are an argument against DNA-centrism. This is because all cells in our body have the same genome. If the fertilized egg (zygote) has a mutation, all cells in our body necessarily have that mutation. Yet, genetic diseases tend to affect specific organs in our body: blood, brain, muscles, heart, lungs, intestines, eyes, ears, etc. How could that be if all cells harbour exactly the same genome? Answer: it is the difference in expression of genes. Do genes express themselves? Of course not. Factors outside DNA trigger gene expression. Additionally, genetic diseases often start at different ages. For example, symptoms of Huntington's Disease typically appear in middle-aged people. How could that be? The genome doesn't change with age. Again: gene expression changes with age, not the gene itself. This is an argument against DNA-centrism because these examples show that DNA is passive, and factors outside DNA cause gene expression in specific organs at specific times. To understand this correctly, the whole cell should be in the centre ('cell-centric view of life').
Origin of Life
The DNA-centric view of life spectacularly fails in the context of the Origin of Life. That's a hint that should make us think again. Life didn't and couldn't start with DNA. That is because DNA is a dead and meaningless molecule. It has no activity on its own. That is the famous vicious circle: the enzymes that transcribe and translate DNA must be present before those very enzymes can be produced.
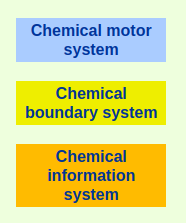
Another powerful reason why DNA-centrism is wrong is that DNA is only one of the three components that constitute life:
|
| Tibor Gánti model of life [3] |
Not one of the subsystems is dominant, all three determine the living organism. One subsystem is a chemical system and nothing more.
This blog is an attempt to summarize my position, not a review of the literature. My position does not downplay the importance of DNA [4], I emphasize the passive role of DNA. This does not contradict any facts. However, my position appears non-mainstream due to sloppy language use in the scientific literature [5].
Acknowledgements
Susan checked my English. Thanks!
|
| random DNA (source) |
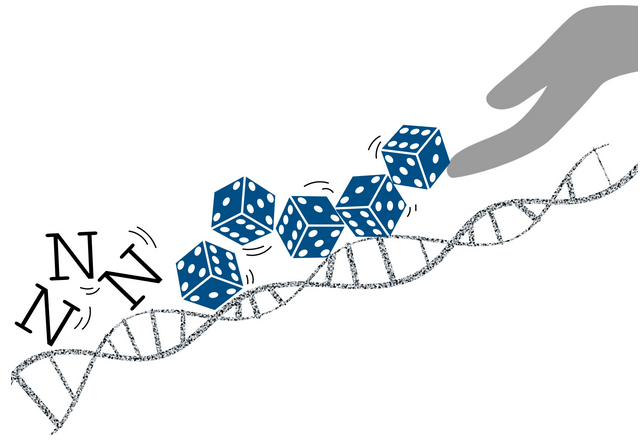
Can a random DNA sequence contain a gene by pure chance? If a 'gene' is defined as a sequence consisting of a number of bases that is divisible by three, starts with a START codon and ends with a STOP codon, then simple statistics show that genes can be found in random DNA. But, do random genes exist in reality?
Yes, short 'genes' can easily be found in random DNA by pure chance if the random sequence is long enough. The presence of START and STOP codons determines the length of the gene. START and STOP codons are triplets and consist of three bases (nucleotides). Everything between the START and STOP must consist of triplet codons. A STOP codon terminates the synthesis of each protein. The distance between a START and a STOP codon equals the length of a potential protein-coding gene. If that length is short, the corresponding protein will be too short to be useful. The START and STOP codons occur by chance in a random sequence. Therefore, the gene length is distributed according to statistical laws. The length of a gene is important. The statistics show that the maximum gene length found in a string of random DNA of 50 million bases is 354 base pairs = 118 triplet codons. See figure above. If transcribed and translated, those sequences would produce a protein of 118 amino acids in length. In the above simulation, most of the hypothetical genes are less than 150 base pairs or 50 amino acids in length. Proteins smaller than 50 amino acids are called peptides. A number of peptides are useful, but most functional proteins in animals and plants are much longer: on average 486 amino acids.
In the above computer simulation, only one STOP codon is used for simplicity. That's why mainly peptides are found. If all 3 STOP codons are used in the simulation, ORFs are almost twice as short [2]. On the other hand, if a random sequence would have the length of the human genome, that is 3.5 billion base pairs or 60 times bigger than in the above simulation, than the maximum length of the hypothetical genes would be 470 bp. That is 1.35x longer [2].
In hindsight, it is not surprising that 'genes' can be found in random DNA. The beauty of the genetic code is that any 3-letter combination of A, T, C, G is a valid codon, including START and STOP codons. That means, that any such a sequence is potentially a protein-coding gene. The similarity between real and random genes is that they consist of a sequence of the same 4 letters A, T, C, G. On that level they cannot be discriminated. The differences lie elsewhere (see below).
Interestingly, from the above computer simulation, several mathematical laws emerge.
Mathematicians call the graph a Power Law. Please keep in mind that all this is purely mathematics. It depends on the underlying assumptions whether the results apply to living organisms. For now, it seems that genes of useful length could be found in random DNA with the length of the human genome (3.5 billion base pairs).
But does this sort of mathematics apply to the real world? Does random DNA exist in the real world? Yes, it could be. Our genome consists of 90% 'junk'. Junk DNA is not pure random DNA, it has an evolutionary history. It is subject to random mutation and decay. Therefore, it approaches random DNA. So, there are ample opportunities to find genes in junk DNA. Curious genome researchers have searched for functional random genes in real genomes. But first: how do you detect those random genes? How do you discriminate between 'normal' genes and 'random' genes? Again, both are a series of 4 letters in a sequence. Random sequence genes must be novel genes or de novo genes. They are also called orphan genes for obvious reasons. Since they are created from scratch, they do not have identical or similar genes in the genomes of closely related species. Furthermore, the sequence of the putative gene must be present in non-coding DNA of closely related species in the same location on the chromosome. A researcher concluded:
"Although considered an extremely unlikely event, many genes emerge from previously noncoding genomic regions. (..) De novo genes arise from previously noncoding DNA, are short, and are expressed at low levels. (...) While most of the de novo genes are lost, a fraction of them becomes essential. [3]
Intriguingly, the ORFs of de novo genes are shorter than those of evolutionary old genes, but longer than expected by chance according to this researcher. We have seen in the above computer simulation that random genes tend to be smaller than real genes. Even more surprising, de novo genes can become essential.
In humans, at least three human protein-coding genes have emerged since the divergence with chimps some 7 million years ago. These loci are noncoding DNA in other primates (that is part of the definition of random genes). Other studies estimate that 18 such cases are present in a genome of 24,000 protein-coding genes [4]. That's interesting, but the problem with this method is that the noncoding sequences could be the modified descendants of ancient genes or pseudo-genes. So, we cannot be 100% sure that they are truly random.
In 2013 genome researcher Sean R. Eddy proposed the Random Genome Project:
"Suppose we put a few million bases of entirely random synthetic DNA into a human cell. (...). Will it be reproducibly transcribed into mRNA-like transcripts, reproducibly bound by DNA-binding proteins, and reproducibly wrapped around histones marked by specific chromatin modifications? I think yes." [1]
Exactly this experiment has been done recently [5]. Investigators inserted a synthetic DNA sequence in yeast and in mouse cells. In yeast cells, the DNA was being read (transcriptional activity), but the mouse cells did nothing. The researchers concluded that regulatory DNA elements are required in order for the gene to be read. But reading a gene is only the first step. The product (mRNA) must be transported to the cytoplasm and translated by ribosomal machinery and transfer-RNA (tRNA) to a protein. Furthermore, the resulting protein must be able to fold in to a functional 3D form. A further requirement is that the new protein must fit into the existing network of biochemical reactions of the cell and the organism. Of course, it does matter whether the gene is active in every cell of the organism, or just in specific tissues or organs. Also, in what quantities it is produced. And whether it is produced in the embryo or in the adult, in males or in females. A lot of requirements indeed. No wonder that there aren't a lot of genes that originate from random DNA.
The discussion so far is about the origin of new genes from random DNA in real genomes of real organisms belonging to real species. But ultimately, the very first genes on Earth must have a random origin. How else could they originate? They have no ancestors. One genome researcher, Senapathy [6], did computer simulations like the simulations discussed here and has put forward a theory that long ago animals and plants originated in primordial ponds from random DNA genomes. For a few thousand reasons, genomes cannot be created from random DNA fragments ('genes'), and organisms cannot be created from random genomes. The ultimate reason is DNA- or genome centrism: the idea that a genome can create an organism. My next blog will be about 'DNA-centrism'.
Rolie Barth contributed the statistical analysis, corrected the draft and made suggestions for improvement. Susan checked my English. Thanks both!
De schilder die ons huis aan het schilderen was, merkte op dat de kitlaag van de ramen aangetast was. Eerlijk gezegd was het mij nog niet opgevallen. Het kwam door slakken zei hij. Hij raadde dubbelzijdig plakband aan. Maar ik vond het nogal bizar dat slakken een (giftige?) kunststof zouden eten. Er is toch voedsel genoeg in de tuin?
Ik wilde eerst weten of het hele verhaal klopte. Ik had nooit slakken op de kitrand gezien. Dat zou dan 's nachts moeten gebeuren? Er was maar één manier om het raadsel op te lossen: die slakken op heterdaad betrappen. Ik wilde 's nachts de Seissiger wildcamera opstellen. Toen ik in het donker de tuin in liep, hoorde ik gekraak onder mijn voeten. En ja hoor: ik trapte op een slak. LED-lampje er bij gehaald: het wemelde van de slakken op het pad. Gefotografeerd en ingevoerd in waarneming: bijna allemaal Segrijnslakken. Ze hebben een grotere schelp dan de bekende huisjesslakken en de schelp heeft allerlei patronen en is fraai gekleurd. De soort komt van oorsprong uit Zuid-Europa. Volgens waarneming.nl heeft de soort inmiddels de status 'algemeen' en 'inheems' en dus niet 'exoot'. Ze eten vooral tuinplanten en zouden zelf ook goed te consumeren zijn [1]. Dat laatste was ik niet van plan.
Dit zijn de slakken die ik 's avonds tegen tienen aantrof op het tuinpad:

|
| Segrijnslak 17 sep 21:36 FP5 ©GK |

|
| Segrijnslak 17 sep 21:37 FP5 ©GK |

|
| Segrijnslak 17 sep 21:38 FP5 ©GK |

|
| Segrijnslak 17 sep 21:39 FP5 ©GK |

|
| Zwartgerande tuinslak 17 sep 21:40 FP5 ©GK |
Maar goed: 's avonds slakken op het tuinpad is nog geen bewijs dat ze ook van de kitlaag eten. Ik heb de wildcamera op timelapse gezet: iedere 5 min een opname. Op de sensor zetten werkt niet voor deze kleine en langzame dieren. En inderdaad, van 00:35 tot 03:30 u., was er een slak actief op de kitrand. Op heterdaad betrapt! Zo'n 3 uur op dezelfde plek!
|
|
|
Seissiger wildcamera 19 sep 2025 00:30 Rode pijl: slak op de crime scene. Groene pijlen: 2 aangetaste plekken. |
Uit de opnames blijkt dat de slak naar een nieuwe plek is gekropen:
|
|
|
Seissiger wildcamera 19 sep 2025 00:35 Op heterdaad betrapt. Rode pijl: slak. Groene pijlen: 2 aangetaste plekken |
Nadat hij zijn buikje vol gegeten had met de overheerlijke kit, verdween hij weer. De volgende ochtend was er een derde aantastingsplek te zien op de plek waar de slak bezig was geweest. Ik acht het voor 100% bewezen dat slakken de kit opeten. En voor 99% zeker dat het de Segrijnslak was. Omdat het donker was en de opnames in zwart-wit zijn, kan ik niet met 100% zeker concluderen dat het om deze soort gaat. Als je het 100% zeker wilt weten, zou je het DNA in de uitwerpselen moeten analyseren.
De vraatsporen zijn vaak geribbeld en ongeveer 1 cm lang. Vaak vind je een langwerpig uitwerpsel in de buurt. Dat is een hint.

|
|
Zo herken je de slak: schade beeld in de kit met uitwerpsel |
Segrijnslak in zijn natuurlijke milieu:
|
|
| Segrijnslak op klimhortensia (SONY A6700 +macro) |
|
|
| detail van de kop |
Gebeld met het schildersbedrijf. Ze stuurden vrij snel een glaszetter die de kitlaag begane grond verving door een ander product die de slakken niet zouden lusten. Ieder zijn vak. De kit is nu een week oud zonder schade.

|
| Jika Sikaflex beglazingskit. |
Aan de voorkant van het huis was de kit nog niet vervangen en ik betrapte een 100% Segrijnslak op heterdaad op klaarlichte dag!
|
|
| 100% Segrijnslak Cornu aspersum 7 Okt 2025 13:40u |
Hij was op dat moment 'in rust', maar hij zat op een aangetaste plek op de kit. Het is nu 100% bewezen dat het Segrijnslakken de kit aantasten. Ik heb hem voorzichtig los gemaakt en in het bos gezet. Ik heb nog geen gewone huisjesslakken dit zien doen.

|
Hij was actief aan het kruipen toen ik hem naar binnen haalde voor de foto. Bij wijze van experiment heb ik hem op de nieuwe slakken-bestendige kit gezet. Hij had geen belangstelling, hij kroop gewoon weg. Het kan zijn dat al zijn buikje vol had...
Segrijnslak (wikipedia):

|
| Wespspin ♀ 27 aug 2025 Sony A6700 + 90mm macro ©GK |
Het gebeurt zelden dat een vrouwelijk fotomodel zich vrijwillig aanbiedt. Zeker niet om vervolgens 2 dagen en nachten stil te blijven zitten. En al helemaal niet dat ze toestaat om haar heel dicht te benaderen.
Dit vrouwtje Wespspin bleek een ideaal fotomodel. Ze is exotisch mooi. Ze had zelf voor de ideale witte achtergrond gezorgd. Op ooghoogte. Perfecte keuze. Normaal bouwen wespspinnen hun web dicht bij de grond tussen het gras. Dan zijn ze moeilijk te fotograferen. Vandaar dat ik zo blij was met dit vrouwtje. Het is uitzonderlijk dat je macrofoto's kunt maken van een levende spin. Ik had nooit eerder een wespspin gezien. Dit was mijn eerste.
De Wespspin Argiope bruennichi (Wasp Spider) is een spin afkomstig uit Zuid-Europa. De soort wordt aangetroffen in graslanden die zelden gemaaid worden maar ook in heidevelden. Vandaar dat het belangrijk is voor natuurbeheerders om dit soort graslanden niet te maaien!

|
| Wespspin 28 aug 2025 Sony A6700 90 mm macro f/14,0 1/125 ISO400 ©GK |
|
|
| Wespspin 28 aug 2025 Sony A6700 90 mm macro f/8,0 1/125 ISO100 ©GK |
De spin heeft 2 primaire ogen en 4 extra secundaire ogen. De primaire ogen zijn naar voren gericht. Van de extra ogen is de linker naar links, en de rechter naar rechts gericht. Zij moet mij en de camera dus uitstekend gezien hebben! En toch blijft ze stil zitten. Wonderlijk! Hoewel, als je er over nadenkt, als er iemand een geduldig karakter zou moeten hebben, dan is het wel een spin. Hartelijk dank voor je geduld! Je bent het beste fotomodel dat ik ooit gehad heb!
De wespspin richt zich vooral op springende en laagvliegende prooien zoals sprinkhanen, libellen en kevers, die tussen de grassen leven. Ik heb inderdaad in het hoge gras rondom het huisje een Grote groene sabelsprinkhaan, een Bruinrode heidelibel en een Hennepnetelgoudhaan (is een kever) gezien. Dat zou dus allemaal kunnen kloppen. We hebben hier te maken met een klein deel van een ecosysteem!

|
| Grote groene sabelsprinkhaan ©GK |

|
| Bruinrode heidelibel ©GK |

|
| Hennepnetelgoudhaan ©GK |
Rolie Barth maakte mij erop attent dat deze wespspin maar 5 poten heeft! Laat ik dat nu totaal 100% over het hoofd gezien hebben! Ik was zo gefocust op een goede foto maken dat ik niet gezien heb dat er 3 poten ontbraken. Een blunder. We hebben hier dus te maken met een beschadigd vrouwtje Wespspin. Ik weet niet of dat iets te maken heeft met haar lethargisch gedrag.
Ik zag op de onderkant van een blad van een kiemplantje een groepje van 28 eitjes. Intrigerend. Een heel regelmatig patroon. We hebben een tuin vol planten en uitgerekend op dit potplantje worden eitjes afgezet. Van welk dier zijn ze afkomstig?
Aan de vinger zie je hoe klein de eitjes zijn. Ik heb de foto ingevoerd in
waarneming.nl en deze werd even later handmatig goedgekeurd door de
'wantsen-moderator' als:
Groene schildwants. Dat is een algemene soort schildwants. Maar, een andere foto van
hetzelfde groepje eitjes werd door een 'vlinder-moderator'
automatisch goedgekeurd als
Groot Koolwitje! Het kan toch niet zo zijn dat ze goedgekeurd worden als twee verschillende
soorten!
Het is wel zo dat de eitjes enigszins op elkaar lijken, maar dat experts tot
strijdige oordelen komen is nieuw voor mij. Voorlopig accepteer ik de 'Groen
schildwants' want die is handmatig door een expert goedgekeurd. Dit
terzijde, want dit blog gaat over dat wonderbaarlijke cluster eitjes en wat
er van terecht is gekomen.

|
| 5 juli 2025 |
Twee dagen later zijn er allemaal zwarte figuurtjes, een soort emoji, en 2 rode stippen te zien. Bizar! Het lijken wel pingpongballen met merktekens. Het moeten de eerste zichtbare tekenen van het embryo zijn.

|
| 5 juli 2025 |
Van dichtbij zie je met de juiste belichting regelmatige kleine uitsteekseltjes op de randen van de eitjes.

|
| 6 juli 2025 |

|
| 8 juli 2025 |
Op de vijfde dag zie ik plotseling kleine wantsen. Maar ze blijven op het
ei cluster zitten. Soms bewegen ze een heel klein beetje. Het zijn niet de
volwassen dieren.

|
| één groen individu... (8 juli 2025) |
Tussen alle zwarte zit er een groene met felrode oogjes. Die ogen komen
overeen met de twee rode stippen in het ei. Is dit groene individu een
volgende stadium?

|
| 10 juli 2025 |

|
| 15 juli 2025 |

|
| 15 juli 2025 |
Een ander individu is op de tafel gevallen en weer met die komische omhoog
gerichte antennes. Het zijn een soort miniatuur robotjes.
. . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . .
GGG04358.JPG)
|
| 24 juli 2025 |
De volgende dag zijn ze plotseling allemaal verdwenen. Jammer! Nu ben ik ze kwijt. Totdat ik een week
later een verdwaald volwassen exemplaar op de witte tegels van de WC zie zitten. Dit exempaar zou afkomstig kunnen zijn van het groepje eieren van 3
juli. Maar ik weet het niet natuurlijk niet zeker.
GGG04358detail.JPG)
|
|
Let op de uitstekende rode ogen en de eindeloos veel kleine zwarte stipjes op het schild. |

|
| 25 jul 2025 |
De volgende dag vind ik een volwassen exemplaar, herkenbaar aan die bruine
vlek op achterlijf. Dat zijn de punten van de vleugels. Het heeft een
perfecte schutkleur op een groen blad. Die zie je makkelijk over het hoofd
als je er niet naar op zoek bent.
De Groene schildwants is niet zeldzaam. Het is volgens waarneming één van de algemeenste grote wantsen in Nederland. Maar het is wel zeldzaam dat je de hele reeks van ei tot volwassen dier kunt volgen op een Rode Zonnehoed plantje dat je in een bloempot hebt opgekweekt uit een zaadje. Het hele verhaal vond ik de moeite waard om over te bloggen.
Wikipedia (NL) heeft een heel uitgebreide beschrijving van de Groene schildwants. Dat is best bijzonder voor wikipedia.

|
| Pyjamaschildwants Graphosoma italicum 19 juli 2025 |
Dat is nu weer het tegenovergestelde van een schutkleur! Waarom? Wat is de bedoeling van die opvallende rode kleur?
Tenslotte had ik vorig jaar ook nog een andere wants:

|
| Roodpootschildwants 4 sept 2024 |
zat zomaar uit het niets op een vinger! Ook niet handig van zo'n beestje, tenminste als je niet opgegeten wilt worden. Maar wel handig voor de fotograaf.
De onderstaande wants was volkomen 'gratis':

|
| Meidoorn-kiel-wants 08 aug 2025 |
In 2021 had ik nog een prachtige schildwants:

|
| Bessenschildwants 24 aug 2021 |
In totaal heb ik 9 schildwantsen op mijn soortenlijst. Maar bovenstaande
zijn wel de mooiste. Dat was het weer.